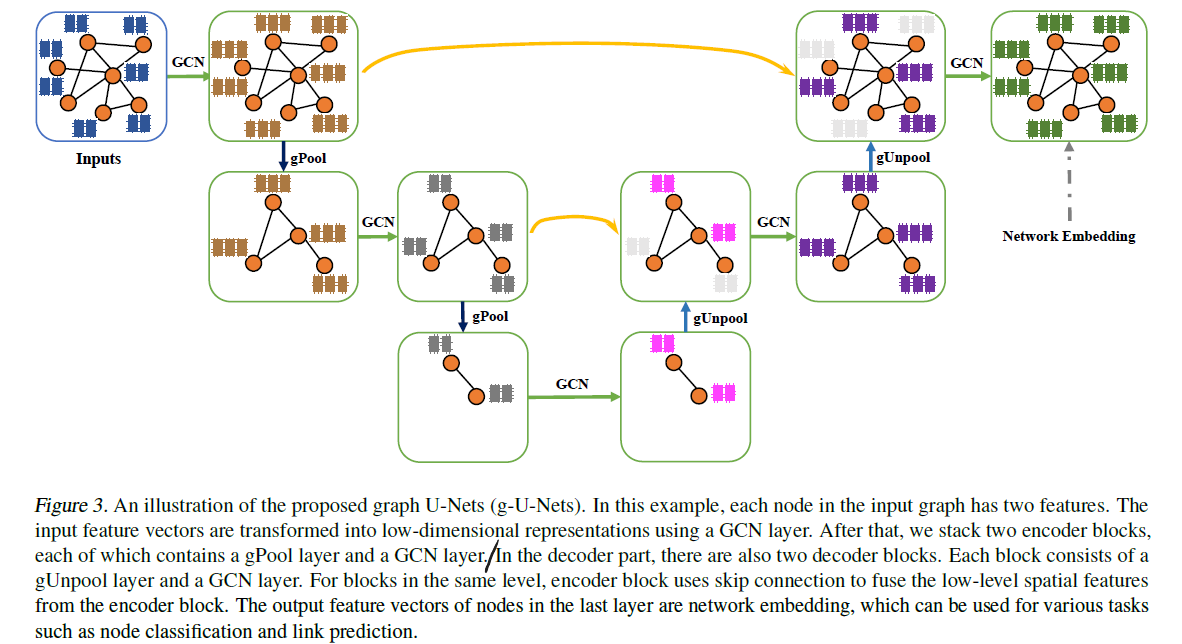
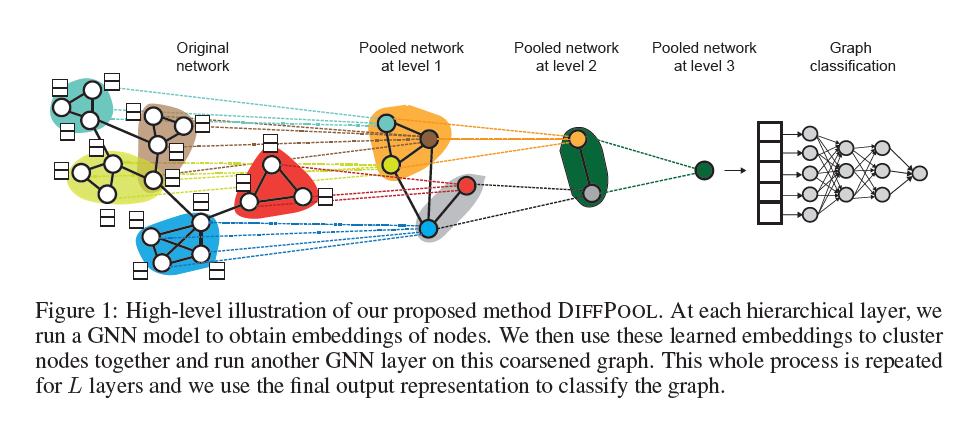
이번 포스팅에서는 Apache Spark의 기본에 대해 정리해본다. 평소에 자주 사용하지만 정확히 숙지하지 못한 부분들을 정리하는 데에 목적이 있다.
Apache Spark 기본
1. 기본 개념
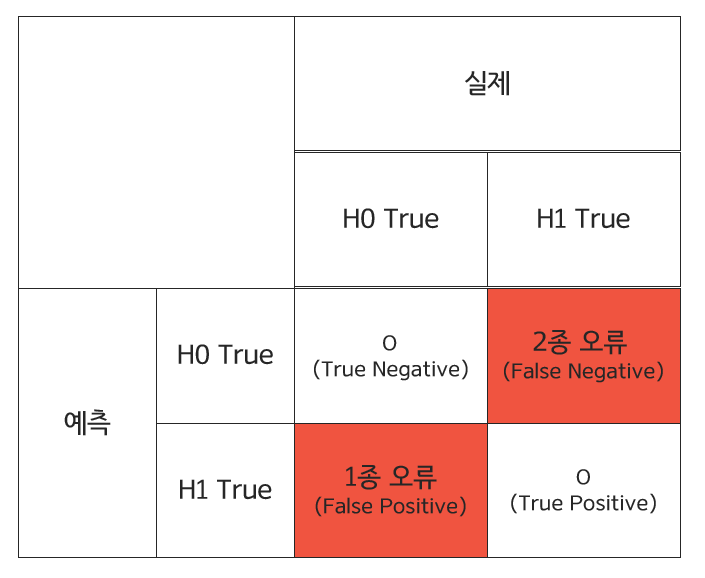
Spark는 여러 컴퓨터를 모은 클러스터를 관리하는 framework다. 이 때 클러스터는 Hadoop Yarn, Mesos 같은 클러스터 매니저가 관리하게 되며, Spark는 클러스터의 데이터 처리 작업을 관리 및 조율하게 된다.
사용자는 이 클러스터 매니저에 애플리케이션을 제출하고, 클러스터 매니저는 애플리케이션 실행에 필요한 자원을 할당하게 된다.
Spark 애플리케이션은 Driver 프로세스와 다수의 Executor 프로세스로 구성된다. 이 때 Driver 프로세스는 메인 함수를 실행하고, 전반적인 Executor 프로세스의 작업과 관련된 분석, 배포 및 스케쥴링을 담당하게 된다. Executor 프로세스는 Driver 프로세스가 할당한 작업을 수행한 후 다시 보고한다.
즉, 이름에서도 알 수 있듯이 Driver 프로세스는 계획을 수립하고 명령을 내리는 컨트롤 타워의 역할을 하고, Executor 프로세스는 이를 수행하는 일꾼의 역할을 한다는 것을 알 수 있다.
아래 그림은 클러스터 매니저가 물리적 머신을 관리하고 스파크 애플리케이션에 자원을 할당하는 방법을 나타낸다. 사용자는 각 Executor에 할당할 노드 수를 지정할 수 있다.
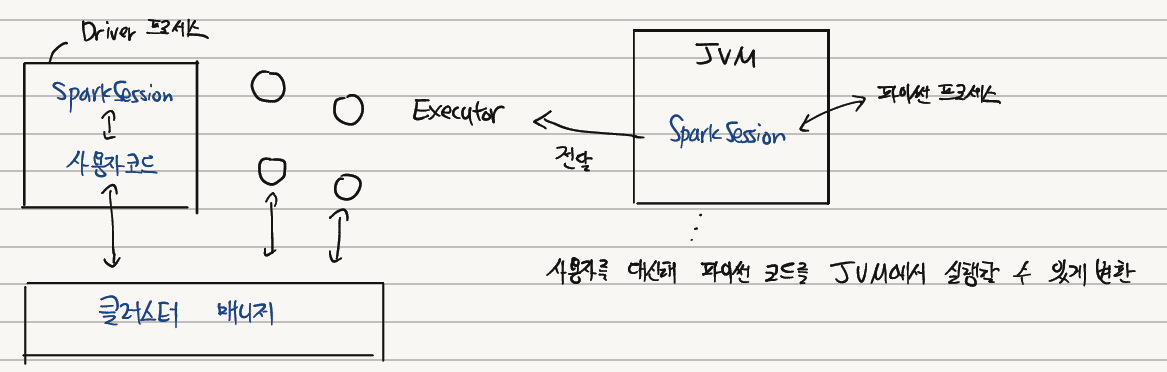
Spark는 파이썬, 자바, 스칼라, R을 지원한다. 예를 들어 파이썬을 사용한다고 하면 Spark는 사용자를 대신하여 파이썬으로 작성한 코드를 Executor의 JVM에서 실행할 수 있는 코드로 변환한다. 이 과정에도 역시 비용이 든다. 따라서 만약 사용자가 큰 비용을 수반하는 UDF를 생성해야 한다면 스칼라나 자바로 이를 구현하는 것이 좀 더 효율적이다.
SparkSession은 앞서 기술한 Driver 프로세스이다. 사용자가 정의한 처리 명령을 클러스터에서 실행하게 되는데, 하나의 SparkSession은 하나의 Spark 애플리케이션에 대응한다.
그리고 SparkSession의 SparkContext는 클러스터에 대한 연결을 나타낸다. 이 SparkContext를 통해 RDD 같은 저수준 API를 사용할 수 있다.
Spark에는 3가지의 구조적 API가 존재한다. DataFrame, Dataset, SparkSQL이 그 대상들이다. 다만 Dataset의 경우 파이썬이나 R에서는 지원되지 않는다. 이들 개념에 대해서는 다음 장에서 살펴본다. Spark DataFrame이나 Dataset은 수 많은 컴퓨터에 분산 저장된다.
파티션은 모든 Executor가 병렬로 작업을 수행할 수 있도록 chunk로 데이터를 분할한 것이다. 파티션은 또한 데이터가 클러스터에서 물리적으로 분산되는 방식을 나타내고, 만약 파티션이 1개면 Executor가 많아도 병렬성은 1이 된다.
기본적으로 DataFrame과 같은 Spark의 핵심 구조는 immutable하고, 변경하고 싶다면 명령을 내려줘야 한다. 이 명령을 Transformation이고, 이 개념은 Spark에서 비즈니스 로직을 표현하는 핵심 개념이다.
Transformation에는 다음과 같이 2종류가 존재하고, Wide Dependency를 가지는 Transformation은 Shuffle 비용을 발생시키기 때문에 주의가 필요하다.

이렇게 만든 Transformation은 바로 실행되는 것은 아니다. 왜냐하면 Spark는 Lazy Execution이라는 규칙을 갖고 있기 때문이다. Spark는 특정 연산 명령이 내려지면 데이터를 즉시 수정하는 것이 아니라 실행 계획을 생성하고, 마지막 순간 전에 원형 Transformation을 간결한 물리적 실행 계획으로 컴파일한다. 그리고 이 과정 속에서 Spark는 사용자가 쉽게 할 수 없는 최적화를 수행하여 작업의 효율성을 높인다.
Action은 실제 연산을 지시하는 명령이다. 예를 들어 count 명령은 DataFrame의 전체 레코드 수를 반환하는 Action이다. 이렇게 Action을 지정하면 Spark Job이 시작된다. 참고로 Spark 애플리케이션은 1개 이상의 Job으로 구성된다.
2. 구조적 API
구조적 API는 데이터 흐름을 정의하는 기본 추상화 개념이고 앞서 언급하였듯이 DataFrame, Dataset, SparkSQL로 구성된다. 구조적 API는 비정형 로그파일부터 정형적인 Parquet 파일까지 다양한 유형의 데이터를 처리할 수 있다.
DataFrame과 Dataset의 공통적인 특성은 다음과 같다.
- 잘 정의된 Row, Column을 갖는 분산 테이블 형태의 컬렉션
- 결과를 생성하기 위해 어떤 데이터에 어떤 연산을 적용해야 하는지 정의하는 지연 연산의 실행 계획
- 불변성을 지님
- DataFrame에 액션을 호출하면 Spark는 Transformation을 실제로 실행하고 결과를 반환함
- DataFrame을 사용하면 Spark의 최적화된 내부 포맷을 이용할 수 있음
마지막 부분이 중요한데, 사실 Spark는 사용자 모르게 (물론 살펴보면 알 수 있다.) 복잡한 연산 과정에 대해 수많은 최적화를 수행한다. 이 때 구조적 API인 DataFrame을 사용하면 Spark의 최적화 과정은 더욱 빛을 발하게 된다. 이는 결국 정확히 어떤 작업을 하는지 인지하고 있을 때만 저수준 API인 RDD를 호출해야 하며, 명확한 목적과 설계가 잡혀있지 않는 이상 웬만하면 DataFrame 수준에서 작업을 진행하는 것이 좋다는 뜻이다.
구조적 API의 실행 과정에 대해 살펴보자.
일단 구조적 API를 이용하여 코드를 작성한다. Spark는 이를 논리적 실행 계획으로 변환한다. 이후 논리적 실행 계획을 물리적 실행 계획으로 변환하며 이 과정에서 최적화를 할 수 있는지 확인한다. 최적화는 Catalyst Optimzer에 의해 이루어진다. Spark는 알아서 실행 과정 속에서 최적화를 해주는 것이다!
이후 Spark는 클러스터에서 물리적 실행 계획, 즉 RDD 처리를 실행한다. 물리적 실행 계획은 일련의 RDD와 Transformation으로 변환되는데, Spark는 구조적 API로 정의된 쿼리를 RDD Transformation으로 컴파일한다. 이 과정 때문에 Spark는 컴파일러로 불린다.
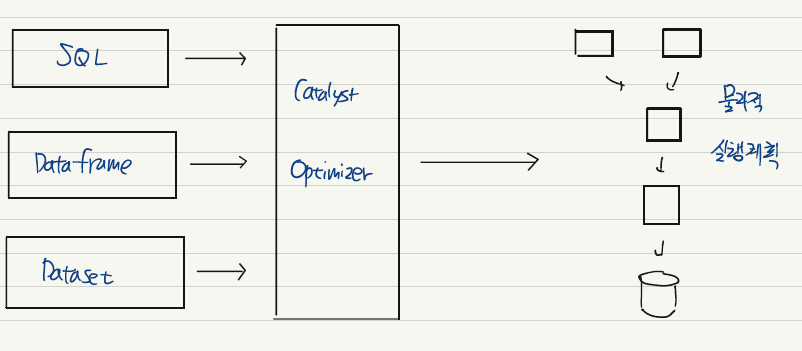
사용자는 DataFrame과 같은 구조적 API에 기반하여 코드를 짜게 될 것이다. 그렇게 하면 이 추상화된 개념을 놓고 Spark는 실제 실행 계획을 수립하면서 최적화를 하고 최종적으로 결과물을 반환하게 된다.
3. 저수준 API
Spark에서는 2가지 저수준 API를 지원한다. 하나는 앞선 장에서 언급한 RDD이다. 다른 하나는
Broadcast 변수와 Accumulator와 같은 분산형 공유 변수를 배포하고 다루기 위한 API이다.
3.1. RDD
RDD는 resilient distributed dataset의 약자로, 다수의 서버에 걸쳐 분산(distributed) 방식으로 저장된 데이터 요소들의 집합을 의미하며, 병렬 처리가 가능하고 장애가 발생해도 스스로 복구 가능(resilient)하다.
Dataset과 DataFrame이 존재하기 전에는 RDD 자체로 많이 사용하였지만 현재는 이 자체로는 특수한 목적 외에는 잘 사용되지는 않는다. 물론 사용자가 작성한 Dataset, DataFrame 코드는 앞서 기술하였듯이 실제로는 RDD로 컴파일되어 수행된다. RDD를 사용하면 Spark의 여러 최적화 기법을 사용할 수 없기 때문에 세부적인 물리적 제어가 필요할 때만 RDD를 명시적으로 사용해야 한다.
3.2. 분산형 공유 변수
브로드캐스트 변수는 불변의 값을 closure 함수의 변수로 캡슐화하지 않고 클러스터에서 효율적으로 공유하게 해준다. 모든 워커 노드에 큰 값을 저장하여 재전송 없이 많은 Spark 액션에서 재사용이 가능하다. 모든 Task마다 직렬화할 필요 없이 클러스터의 모든 머신에 캐시하게 된다.
브로드캐스트 변수는 다방면에서 활용될 수 있는데, 이후에 설명할 broadcast join에서 그 효과를 직관적으로 이해할 수 있을 것이다.
어큐멀레이터는 Spark 클러스터에서 row단위로 안전하게 값을 갱신할 수 있는 변경 가능한 변수를 제공한다. 이를 디버깅용이나 저수준 집계용으로 사용할 수 있다. 예를 들어 파티션 별로 특정 변수의 값을 추적하는 용도로 사용할 수 있고 병렬 처리 과정에서 더욱 효율적으로 사용할 수 있음. 기본적으로 Spark는 수치형 어큐멀레이터를 지원하나 사용자 정의 형태도 가능하다.
어큐멀레이터에 이름을 지정하면 이 실행 결과는 Spark Web UI에 표시되기 때문에 모니터링하기에 매우 편리하다.
4. 애플리케이션, Job, Stage, Task 개념
이전에 Transformation과 Action의 개념에 대해서 설명하였다. Action이 실제로 어떤 연산을 하는 작업이라고 하였다. 이 Action 하나당 1개의 Job이 존재한다. 그리고 이 Job은 일련의 Stage와 Task들로 구성된다.
Stage는 다수의 머신에서 Task의 그룹이다. Task들은 모두 동일한 연산을 수행하게 되는데, 파티션 1개당 1개의 task가 주어진다. 그리고 Executor는 1개의 파티션에 대해 작업을 처리하게 된다. 그림으로 보면 아래와 같다.
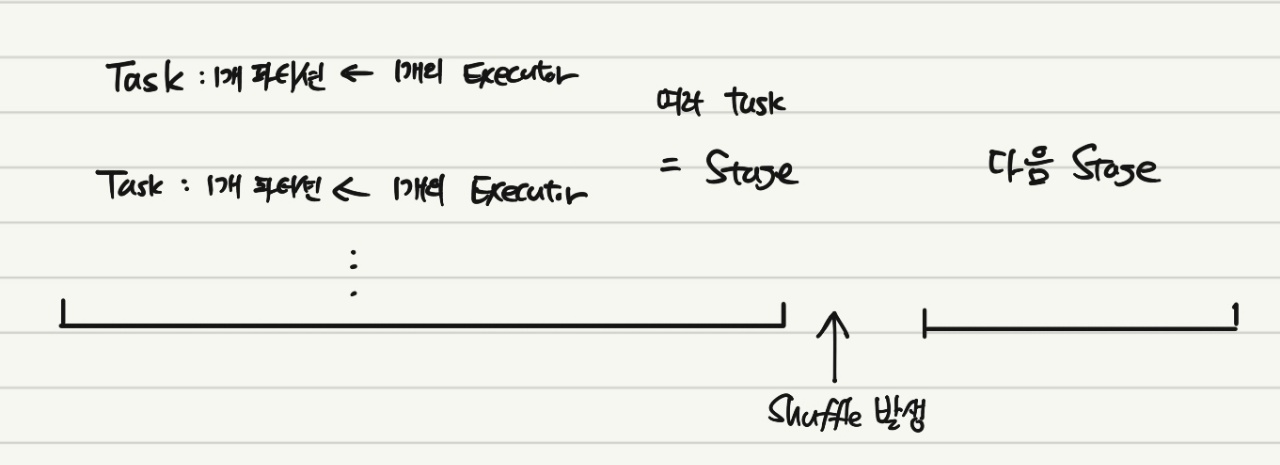
위 설명처럼, 각 Stage는 Shuffle이 발생했는지의 여부에 따라 구분되게 된다. 참고로 Shuffle은 각 노드 사이에 데이터의 이동이 발생하는 것을 의미한다.
그리고 최종적으로 이러한 여러 Job들이 모여 전체 Spark 애플리케이션을 구성한다.
5. Join
Spark에서의 Join을 찾아보면, Shuffle Join, Broadcast Join, Sort Merge Join이 나올 것이다. 그런데 Shuffle Join은 Spark2.3에서부터 Sort Merge Join으로 대체되었다. (설정으로 변경할 수 있다.) Sort Merge Join이 Shuffle Join에 비해 클러스터내 데이터 이동이 더 적다고 알려져 있다.
Sort Merge Join은 사용자가 일반적으로 DataFrame에 join을 행하면 가장 많이 일어나는 join이다. 먼저 파티션을 정렬한 후 이 정렬된 데이터를 병합하면서 join key가 같은 row를 join하게 된다. 먼저 정렬을 하기 때문에 데이터가 심하게 뒤섞여 있거나 skewed되어 있으면 이 비용이 상당히 크다.
Broadcast Join은 작은 테이블을 큰 테이블에 join할 때 사용된다. 작은 테이블을 클러스터 전체 worker node에 복제하고 이를 캐시하여 계속 사용하는 것인데, 한 번 대규모 통신이 발생하긴 하지만 (이 때의 비용은 클 것이다.) 이후 추가적인 통신이 없기 때문에 굉장히 유용하고 실제로 크게 속도를 향상시켜준다. 보통 이러한 상황에서 Spark는 알아서 Broadcast Join으로 계획을 수립한다. (구조적 API를 사용할 때)
Join 수행 시 시간이 너무 오래걸린다고 생각이 되면 아래 Tip들을 참고하면 좋다.
- Join될 파티션들이 최대한 같은 곳에 있어야 한다.
- DataFrame의 데이터가 균등하게 분배되어 있어야 한다. (not skewed)
- 병렬 처리가 이루어지려면 일정한 수의 고유 key가 있어야 한다.
위와 같은 과정에 대해서 좀 더 자세한 설명이 필요하다면 아래 링크를 참조하면 좋다.
6. Spark Execution 최적화
이전에 기술하였듯이 Shuffle이 발생하면 Stage는 새로 생성하고, 각 Stage는 파티션 개수에 따라 여러 Task로 쪼개진다. 이 Task의 수행 시간은 아래와 같이 또 쪼개볼 수 있다.
Scheduler Delay + Deserialization Time + Shuffle Read Time(Optional) + Executor Runtime + Shuffle Write(Optional) + Result Serialization Time + Getting Result Time
Spark Web UI를 켜셔 Job 모니터링을 하면 자주 볼 수 있는 용어들이다.
Scheduler Delay에 대해 알아보자. Spark는 Data Locality에 크게 영향을 받는다. 데이터가 실제 위치한, 로드된 곳이라고 생각하면 되는데 Spark는 이 데이터 전송을 최소화하기 위해 Task를 데이터와 최대한 가깝게 하여 수행하려고 한다. Data Locality는 아래와 같이 5개로 구분된다.
| Priority |
Locality Level |
설명 |
| 1 |
PROCESS_LOCAL |
데이터가 실행되는 코드와 같은 JVM에 있음 |
| 2 |
NODE_LOCAL |
데이터가 같은 node에 있음 |
| 3 |
NO_PREF |
특별히 locality preference가 없는 곳에 데이터가 존재함 |
| 4 |
RACK_LOCAL |
데이터가 같은 Rack이지만 다른 서버에 존재하여 네트워크를 통해 전송이 필요함 |
| 5 |
ANY |
데이터가 같은 RACK에 있지도 않음 |
상위에 있을수록 좋은 것인데, 만약 Data Locality가 PROCESS_LOCAL이라면 Task는 굉장히 빠르게 진행될 것이다. 아래에 있는 레벨일 수록 실제로 Task를 수행할 때까지의 시간이 길어지고, 이를 Scheduler Delay라고 한다. 즉, 네트워크 전송 비용이 그만큼 사용된다는 것이다.
만약 가용 Executor가 Data Locality를 만족하지 못하면 timeout까지 그냥 기다리게 되기 때문에 spark.locality.wait 파라미터를 조정할 수 있다. 더 나은 Locality를 위해 더욱 긴 waiting time을 설정하거나 waiting time을 0으로 바꿔버림으로써 이전 단계들을 건너뛸 수도 있다.
이 Data Locality가 낮은 레벨에 속해있고, Shuffle 대상 데이터의 크기가 크다면 이후 Shuffle Read/Write Time은 크게 증가하게 될 것이다. Executor Run Time은 data read/write time, CPU execution time, Java GC time으로 구성된다.
Task의 수행 시간에 대해서는 알아보았고, 그렇다면 좀 더 빠르게 작업이 진행되도록 튜닝을 하려면 어떻게 해야할까? 이 부분은 Spark 완벽 가이드 책과, IBM 그리고 Databricks의 포스팅을 참고하여 요약 정리한다.
먼저 간접적인 성능 향상 기법에 대해 정리한다. 일단 구조화 API를 적극 사용해야 한다. 이전에도 언급하였듯이 구조적 API를 사용하면 Spark의 여러 장점들을 그대로 사용할 수 있다. RDD의 사용 영역은 최소화하는 것이 좋다. 특히 Python으로 RDD 코드를 실행하면 JVM과 Python 프로세스를 오가는 많은 데이터를 직렬화/역직렬화해야 해서 많은 비용이 수반된다.
그리고 다음은 Data Locality를 확인해보는 것이다. 지금 수행하고 있는 Task에 대한 Data Locality가 과연 최선인지 파악해보아야 한다. 다음으로는 Shuffle 설정이다. 이 부분이 상당히 중요하다. Shuffle은 일반적으로 큰 네트워크 비용을 요구하기 때문에 지양되곤 한다. 불필요한 Shuffle은 당연히 피하는 것이 좋다. 그러나 애초에 Shuffle이 존재하는 이유는 데이터를 재 분배하여 더욱 효율적인 처리를 가능하게 만들기 위해서이다. 즉, 잘만 사용하면 성능 향상을 이끌어낼 수 있다.
Data Skeweness가 발견되었거나 파티션 수가 너무 적으면 Shuffle이 도움이 된다. 일단 특정 파티션에만 데이터가 몰려있으면 그 파티션에서 task를 수행하는 Executor의 부담이 커지기 때문에 다른 Executor들의 작업이 끝나도 전체 Stage가 끝나지 않는 현상이 발생하게 된다. 또 애초에 파티션 수가 너무 적으면 작업을 수행하지 않는 Executor가 발생할 수도 있기 때문에 파티션 수 조정이 도움이 되는 경우가 많다. 예를 들어 기본 파티션의 수가 200개이기 때문에 task가 200개로 쪼개져 있을 때, 가용 node의 수가 130개라고 하면, 모든 node의 작업이 끝난 후에 오직 70개의 node만이 2번째 작업을 시작하게 될 것이다. 이는 분명 효율을 다소 낮추는 요인이 된다.
추가적으로 Shuffle을 수행할 때는 Output 파티션 당 최소 수십 메가바이트의 데이터는 포함되는 것이 좋으며, 애플리케이션이 실행 중에 메모리를 너무 많이 사용하거나 GC collection이 너무 자주 수행되는 것은 아닌지 확인해보는 것이 좋다.
직접적인 성능 향상 기법에 대해 알아보자. Executor 당 할당되는 CPU 코어의 수, 그리고 CPU 코어에 할당되는 task 수의 재조정을 통해 병렬화를 향상시킬 수 있다.
파티션 재분배(repartition)는 앞서 언급하였듯이 Shuffle을 수반하지만 데이터가 클러스터에 균등하게 분배되므로 Job의 전체 실행 단계를 최적화할 수 있다. 그리고 만약 Shuffle 없이 파티션의 수를 줄이고 싶다면 Coalesce 메서드를 통해 동일 노드의 파티션을 하나로 합칠 수 있다. 구조화 API 상태에서 repartition을 수행하면 생각보다 괜찮은 성능 향상을 보이는 경우가 많다.
현재의 파티션 기준을 변경할 수 있는데, 특정 칼럼을 기준으로 바로 설정할 수도 있고 사용자 정의 파티셔닝을 사용할 수도 있다. 사실 이 부분은 직접 반영해본 적은 없는데, 사용자 정의 파티션 함수를 생성한 뒤 이를 파티션의 기준으로 삼을 수 있다고 한다. 잘 제어하면 skewed된 데이터를 균등 분배할 수 있다.
이론적인 부분에 대해서는 정리를 마쳤고, 개인적으로 Spark Web UI에서 자주 모니터링 하는 항목들에 대해 간략히 설명하고 마치도록 하겠다.
| 구분 |
설명 |
| Shuffle Read Size |
Stage의 시작 단계에서 Executor에 있는 read serialized data의 크기 |
| Shuffle Write Size |
Stage의 끝 단계에서 Executor에 있는 written serialized data의 크기 |
| Shuffle Spill Memory |
메모리에 있는 deserialized된 형태의 데이터의 크기 |
| Shuffle Spill Disk |
spill한 후 disk에 있는 serialized된 형태의 데이터의 크기 |
| Peak Execution Memory |
shuffle/aggregation/join 동안 생성된 내부 데이터 구조에 의해 차지하는 메모리 크기 |
Shuffle Read Size부터 유심히 보게 된다. Shuffle이 발생했을 때 얼마나 많은 데이터에 대해 네트워크 전송 비용이 들어가는지 가늠할 수 있기 때문이다. Shuffle Spill Memory/Disk는 전체 task가 끝난 후에 집계되며 언제나 Spill Memory > Spill Disk 관계이다. Peak Executiom Memory는 앞서 설명한 accumulator 변수인데, 이 값은 Task 내에서 생성된 모든 데이터 구조의 peak size의 총합과 거의 일치한다. 따라서 내가 다루고 있는 데이터의 전체 size에 대해 추정해볼 수 있다.
7. Apache Arrow
Apache Arrow는 Spark에서만 쓰이는 라이브러리는 아니지만, Spark에서 대단히 중요한 역할을 한다. in-memory columnar 데이터 포맷으로 JVM과 Python 프로세스 사이의 효율적인 데이터 전송 및 변환을 수행하는데, 메모리 공유를 통해 빠른 변환을 가능하게 한다. 또한 Tensorflow 및 Pytorch와도 고성능 데이터 교환 수단을 지원하기 때문에 만약 Spark 2.3.0 이상의 버전을 사용하고 있다면 거의 필수적으로 사용해야 하는 라이브러리이다. Arrow가 설치되어 있으면 효율적인 Vectorized UDF인 Pandas UDF를 사용할 수 있다. Pandas UDF를 사용하면 직렬화 overhead가 거의 발생하지 않기 때문에 속도를 굉장히 향상시킬 수 있다.
References
1) Spark 완벽 가이드 by 빌 체임버스, 마테이 자하리아
2) 참고 블로그
3) 참고 블로그
4) 참고 Medium글
5) 참고 Medium글
6) IBM Docs